東京大学医科学研究所(東大医科研) 附属ヒトゲノム解析センターならびに日本IBMは6月8日、新型コロナウイルス(SARS-CoV-2)の変異状況のモニタリングならびにウイルスの感染経路同定に活用可能なシステム「HGC SARS-CoV-2 Variant Browser」を開発、運用を開始したことを発表した。
新型コロナウイルスは、流行の長期化に伴い新規のゲノム変異を蓄積し、時に新たな性質を獲得した変異株となり、各国の感染症対策に大きな影響を与えている。一方、こういったゲノム変異情報を活用することで、変異株よりも詳細なレベルで感染者の新型コロナウイルスが「どういった変異を持っており」「いつどの国から流入し」「どのように感染してきたのか」といった情報を得ることができ、そうした情報を元に変異株の拡大状況や懸念するべき新たな変異株の出現を把握することを知ることができるようになってきた。
こうした取り組みを実現するためには迅速に膨大な数の感染者から取得したウイルスゲノム情報を解析する必要があるが、このような膨大な数のウイルスゲノムを迅速に解析して結果を提供する体制が十分に整備できていなかったという。また、ワクチンの接種も開始されているが、ワクチンの機能を回避する変異株の出現も危惧されており、今後の変異株の状況把握を継続して行っていくことは重要視されている。
こうした背景の中、東大医科研附属ヒトゲノム解析センターとIBMは、新型コロナウイルスのゲノム変異状況のモニタリングならびに、ゲノム変異レベルでのウイルスの感染経路を把握できるHGC SARS-CoV-2 Variant Browserを開発することで、ボトルネックを解消し、情報提供を速やかに行うことができる研究体制の確立を目指しまして共同研究を実施。このたび、IBMの基礎研究部門であるIBM ResearchがCOVID-19向けに開発してきたSARS-CoV-2 Variant Annotatorおよび、IBMでデジタル変革を推進するIBM Garageと、公開したSARS-CoV-2 Variant Browserの技術をベースとしたHGC SARS-CoV-2 Variant Browserを開発し、東大医科研附属ヒトゲノム解析センターが運用を開始したという。
特に今回の取り組みでは、国外からの新型コロナウイルスの流入時期や国内の感染経路を把握するための新機能をIBMと協力して実装したとするほか、東大医科研附属ヒトゲノム解析センターでは、新型コロナウイルスをシークエンス解析した生データをHGC SARS-CoV-2 Variant Browserで可視化できるよう解析するためのデータ解析パイプライン「HGC_CovidPipeLine」も合わせて公開したという。
これにより例えば、変異での検索だけでなく、地域や時期などで検索をかけることができ、その検索条件にあった、変異の状況を簡便に俯瞰できるようになるという。
-

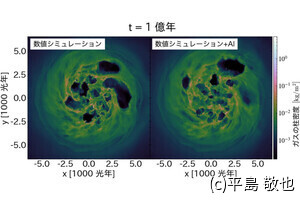
新型コロナウイルスのゲノム変異の俯瞰図(2021年1月以降に日本国内にて報告されたものを活用したという。特定のゲノム変異やLineageなどからも確認することができるとしている) (出所:東大医科研Webサイト)
なお、東大医科研附属ヒトゲノム解析センターでは、今回のシステムをコロナ制圧タスクフォースにおいて収集する新型コロナウイルスゲノムの解析やオリンピック・パラリンピックによる人流に起因するウイル伝播のモニタリングに活用することで、新型コロナウイルスの基礎研究ならびに感染拡大防止策の立案に貢献して行けるのではないかとしている。また、IBMでも、IT技術を活用した取り組みを通じて、引き続き新型コロナへの取り組みを継続する予定だとしている。