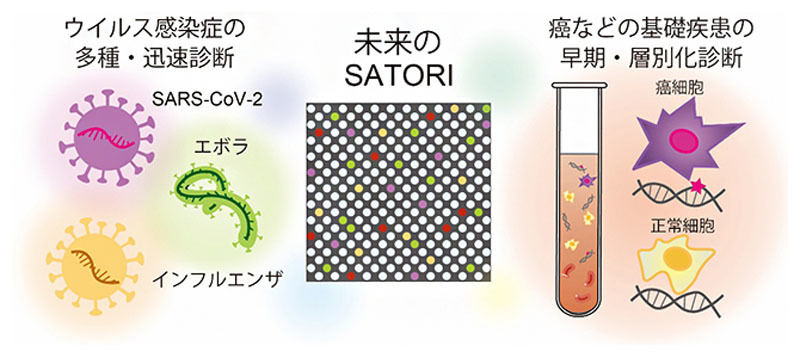
理化学研究所(理研)は4月19日、新型コロナウイルス(SARS-CoV-2)由来のウイルスRNAを「1分子」レベルで識別して5分以内に検出する革新的技術「CRISPR-based amplification-free digital RNA detection(SATORI)法」の開発に成功したことを発表した。
同成果は、理研 開拓研究本部渡邉分子生理学研究室の渡邉力也 主任研究員、同・篠田肇 研究員、東京大学先端科学技術研究センターの西増弘志 教授、同大学大学院理学系研究科の濡木理 教授、京都大学ウイルス・再生医科学研究所の野田岳志 教授らによるもの。詳細は科学雑誌「Communications Biology」(オンライン版)に掲載された。
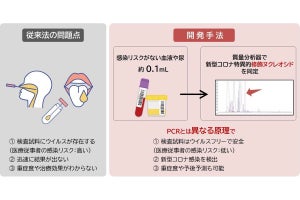
現在、新型コロナウイルス(SARS-CoV-2)の感染診断では、主にタンパク質抗原を検出する方法(抗原検査)とウイルスRNAを増幅して検出する方法(PCR検査)が用いられているが、抗原検査は30分程度でウイルスを検出できるものの、検出感度や特異度の低さに起因する検出精度の問題がある。一方のPCR検査は高い検出感度を有するものの、検出の前処理だけでも最短1時間程度、増幅に起因する検出エラーも発生することから、大量の検出を迅速に解析し、診断につなげることができないという課題があった。そのため、PCRの高い感度と抗原検査の迅速・簡便さを両立するウイルス検出法の開発が求められていた。
今回開発されたSATORI法は、渡邉主任研究員の研究グループが専門とする「マイクロチップを利用した酵素反応の1分子検出技術」と西増教授や濡木研究員の研究グループが専門とする「核酸切断酵素CRISPR-Cas13a(Cas13a)」に関する先進技術を融合させたもので、特定のRNA配列を認識するCas13aと蛍光レポーターの混合液をバイオセンサーとして利用することで、検体中の標的ウイルスRNAの有無を1個ずつ5分以内に識別して検出することが可能だという。
また、検出感度は5fMで、ウイルスRNAの量に直すと1μLあたり約103個。従来の抗原検査法の104~105個/μLと比較すると10~100倍高く、PCR検査の10~102個/μLと比較すると10~100倍低い値だが、SARS-CoV-2感染者の検体中のウイルスRNA量は、103~106個/μLであることから、SARS-CoV-2の感染診断を実施する上では必要な感度を満たしているといえるという。
-
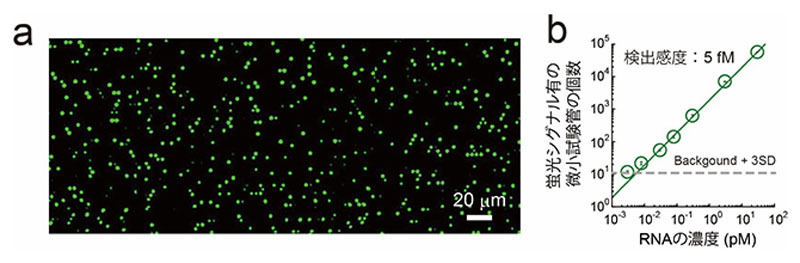
SATORI法によるデジタル検出例。(a)はSATORI法によるマイクロチップの蛍光画像。個々の輝点が1分子のウイルスRNAに相当する。(b)はウイルスRNAの濃度と蛍光シグナルが確認された微小試験管の個数の相関関係。検出感度は5fM。これをウイルスRNAの量にすると、3×103個/μLとなる (出所:理研Webサイト)
なお、SATORI法のランニングコストは約9ドル程度としており、PCR検査の5ドル程度とほぼ同等としているほか、今後、消耗品の大量生産や検出装置の小型化により、安価で素早く多種のウイルス感染症を正確に診断できる次世代の感染症診断法となることが期待できるとしている。