明治大学は、新規に開発したGenomic SELEX法を用いて、大腸菌の持つ約300種類の転写因子のうち200種類以上のゲノム上の結合領域を同定し、転写因子の新分類を提案した。また、ゲノム上の結合領域が一箇所であるSingle-target regulatorsに着目し、その特徴を明らかにした。
同研究は、明治大学農学部農芸化学科の島田友裕専任講師、信州大学基盤研究支援センターの小笠原寛助教、法政大学マイクロ・ナノテクノロジー研究センターの石浜明特任教授らの研究グループによるもので、同研究成果は、2月26日にOxford academicの「Nucleic Acids Research」でオンライン発表された。
ゲノムにある遺伝子のどれが利用されるかは、転写装置であるRNAポリメラーゼおよび環境変化を感知する転写因子の相互作用によって制御されている。これまで、遺伝子個別の転写制御に関しては、関与する転写因子とその作用機序が同定されてきたが、遺伝子を利用する仕組みの全体像については、異なる生物における研究成果や情報を寄せ集めて推測しているのが現状となっている。そのため、ゲノムの全ての遺伝子を対象にした転写制御ネットワークの解明には、新たな研究戦略が求められていた。
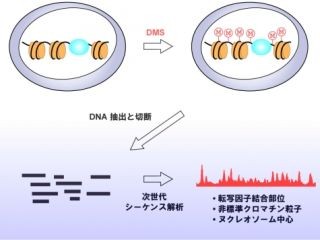
大腸菌の遺伝子は約4,500個あるが、ゲノムの転写制御は約300種類の転写因子とゲノムDNAとの相互作用によって行われている。転写因子の役割によって制御する遺伝子は異なり、ゲノム上の結合領域も異なる。同研究グループでは、そのうちの200種類以上の転写因子を、新規に開発したGenomic SELEX法を用いてゲノム上の結合領域を決定し、それぞれの転写因子が支配する直接的な標的遺伝子群を明らかにした。
また、同研究成果を基盤として転写因子の結合領域数および機能を詳細に解析し、ゲノム上の結合領域数が千箇所前後の転写因子をNucleoid-associated regulators、数百箇所をGlobal regulators、数十箇所をLocal regulators、一箇所であるものをSingle-target regulatorsとして分類することを提案した。さらに、Single-target regulatorsについて、その機能の特徴により、単独結合型、エフェクター存在下活性化型、リン酸化活性化型に小分類することを提案した。
これらの成果から、大腸菌の転写制御ネットワークの全体像が提案された。同研究では、遺伝的背景が等しい単一菌株を利用し、ひとつの研究室で実施された研究成果であることが特徴となっている。同研究は、ひとつの生物で働く全転写因子の制御機能解明、および、生物が遺伝子を利用する仕組みの全体像の理解への先駆けとなることが期待されるということだ。