理化学研究所(理研)は1月28日、フランスなどの研究者らと共同で、「内在性レトロトランスポゾン」と呼ばれる体細胞では発現しないDNA上の繰り返し配列が、発生初期に特異的に活性化することを、マウスの胚を使って突き止めたと発表した。
成果は、理研 オミックス基盤研究領域チームリーダーのピエロ・カルニンチ氏らの研究グループによるもの。研究の詳細な内容は、日本時間1月28日付けで英科学雑誌「Nature Structural & Molecular Biology」オンライン版に掲載された。
内在性レトロトランスポゾンは、ゲノムの中を動くレトロウィルスに類似した配列で、通常の体細胞では発現しない。これまでに、ES細胞や初期の胚で特定の繰り返し配列の発現があることが示唆された例はあるが、網羅的な発現を解析した報告はなかった。
研究グループは、ナノグラムレベルのRNA解析が可能な「nanoCAGE法」を応用したシーケンシングにより、従来なら解析することが難しかった、発生初期の胚におけるレトロトランスポゾン活性の網羅的解析を受精後経時的に行うことにin vivo(生体内)で初めて成功した。その結果、受精によりレトロトランスポゾンが活性化し、発生の進捗と共に転写産物は質的量的に変化することが判明した形だ。
共同研究者であるThe Institute de Genetique et de Biologie Moleculaire et Cellulaire(IGBMC)の研究者らは、ゲノムDNAとタンパク質の複合体であるクロマチンが、胚細胞では、転写が抑制される「ヘテロクロマチン」と呼ばれる凝縮した状態または転写が促進される「ユークロマチン」と呼ばれる緩んだ活性状態のどちらにも属しない「特殊な緩みのある状態」であることを見出し、このクロマチンの特殊な状態が、レトロトランスポゾンの発現を可能にしていると推定。
受精後の卵母細胞で、レトロトランスポゾン由来のRNAが発現していることはすでに知られていたが、今回nanoCAGE法で明らかになったレトロトランスポゾン発現プロファイルは、卵母細胞での発現とは違い、受精後の胚にエピジェネティック(DNAの塩基配列の変化を伴わない)なリプログラミングが起こっていることが示されたのである。
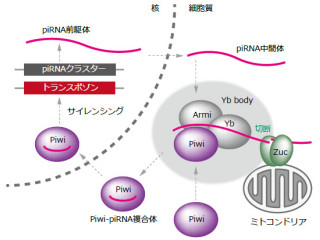
さらに、このレトロトランスポゾン活性を誘導するのは、マイクロRNA(siRNA)よりもさらに短い17塩基のRNAであったことも示された。すなわち、この新しいクラスの短鎖RNAが一連の活性化ループを刺激する因子だったというわけだ。
この新たな知見は、ほ乳類の発生初期の遺伝子発現制御の解明に有益な発見であると、カルニンチチームリーダーらはコメント。レトロトランスポゾンの活性化が、ゲノム上でレトロトランスポゾンが移動するレトロトランスポジションや組み換えをもたらすことで、ゲノム情報の維持を不安定にし、さらには妊娠時の胚のがん化、着床異常をもたらすことが予想されるという。
レトロトランスポゾンの活性化に係るメカニズムをより深く理解することで、不妊に係る腫瘍、欠陥の生成メカニズムの解明が期待されるとしている。