東京大学生産技術研究所、立教大学、みずほ情報総研、国立医薬品食品衛生研究所、神戸大学、海洋研究開発機構、日本電気の7者は、論理的創薬法に適した「医薬品候補化合物(リガンド)」とタンパク質の相互作用解析の高分解能化を可能とする新規手法を開発し、スーパーコンピュータを使って超高速計算することに成功したと発表した。
新規手法を開発したのは、立教大学理学部の望月祐志教授とみずほ情報総研の福澤薫チーフコンサルタント、国立医薬品食品衛生研究所の中野達也室長、神戸大学大学院システム情報科学の田中成典教授らの共同研究グループ。また、海洋研究開発機構のスーパーコンピュータ「地球シミュレータ」を用いて実際の計算を行ったことから、同スパコンを開発したNECとの連携も取られた。
研究の詳細な内容は、3月25日から28日まで開催の日本化学会第92回年会において、「フラグメント分子軌道法によるインフルエンザウイルスノイラミニダーゼと抗ウイルス薬との相互作用解析」(3月27日の講演)として発表された。
今回の開発の経緯は、東京大学生産技術研究所を拠点として行われている「文部科学省次世代IT基盤構築のための研究開発"イノベーション基盤シミュレーションソフトウェアの研究開発"」が基幹にある(2008~2012年度)。その中の研究内容の1つである「バイオ分子相互作用シミュレーターの研究開発」の下で、「革新的シミュレーションソフトウェアの研究開発」および「フラグメント分子軌道法による生体分子計算システムの開発」プロジェクトなどで開発されてきた「ABINIT-MP/BioStation」システムをベースにした、「フラグメント分子軌道(FMO)法」に基づいた創薬におけるプロセスイノベーションに貢献するバイオ分子相互作用シミュレーターの研究開発が行われている。
なお、ABINIT-MPは非経験的FMO法を用いた量子化学計算ソフトウェアで、最新バージョンは4.1。BioStationはバイオ分子相互作用シミュレーターで、こちらの最新バージョンは4.3だ。また、FMO(Fragment Molecular Orbital)法とは、1999年に北浦和夫氏(現・神戸大学特命教授)によって提案された生体分子系に対する効率的な計算手法のことだ。
巨大系を比較的小さなフラグメント(アミノ酸残基など)に分割し、各フラグメントの「モノマー」(重合を行う際の基質のことで、単量体ともいう)と「ダイマー」(2つの同種の分子やサブユニット(モノマー)が物理的・化学的な力でまとまった分子のことをいい、二量体ともいう)の分子軌道計算を並列処理することにより、全系の電子状態をこれまでの手法よりはるかに短時間に高精度で解析することができる近似計算法である(画像1)。
FMO法は計算からはフラグメント間の相互作用エネルギー(IFIEないしPIEと呼ばれる)が得られるため、創薬分野での応用計算が盛んに行われている状況だ。しかし、2体ないし3体のフラグメント展開に基づいた従来手法では、リガンドやアミノ酸の詳細分割を行った場合に、化学的な議論に必要な精度が担保できないという問題があった。
そこで、今回、研究グループは4体(テトラマー)まで展開を拡張した上で「分散力」を取り込める2次摂動論計算に対応した「FMO4-MP2法」を実装して、数値的な精度を確保したのである。FMO4法はフラグメントの展開を4体まで拡張することにより、リガンドの複数分割とアミノ酸残基の主鎖・側鎖分割を伴う高解像度の解析を可能とするアプローチである。今回の実装ではMP2レベルでの計算が可能だ。
また分散力とは、アルキル基やベンゼン環などの疎水性グループ間に働く引力的な相互作用のことだ。共役結合部位間のπ/π型に加え、近年では水素結合に類したCH/π型も認識されている。創薬分野では、リガンドとアミノ酸残基の間の相互作用が特に重要だ。
計算によって分散力を取り込むには、平均場近似を超えて電子相関を導入する必要があり、タンパク質系では2次の「メラー・プレセット摂動論(MP2)」がよく用いられる。MP2計算では、4つの添字を持つ「電子間反発積分」の高速処理が求められるが、ABINIT-MPでは中間ファイルを一切使わない高効率の並列化エンジンを内蔵している仕組みだ。
なお、応分の計算量の増加を処理するため、地球シミュレータのベクトル演算と超並列計算資源を活かしたチューニングも実施された。エイズウイルスの増殖に関わるHIV-1プロテアーゼ(198残基)とロピナビルの複合体のFMO4-MP2計算は、地球シミュレータの128ノードを用いると1.4時間でジョブが完了可能だ。
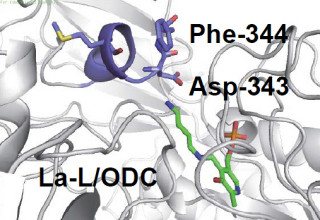
そのほかにも、インフルエンザウイルスの表面に存在し、宿主細胞表面の糖鎖をシアル酸残基の部分で切断する活性を持つ酵素タンパク質のノイラミニダーゼ(NA)とタミフルの相互作用なども計算された(画像2)。ちなみに、インフルエンザウイルスはNAの働きによって、新たに作られたウイルス粒子が感染した細胞から遊離する。そして、タミフルやリレンザなどの医薬品はこの活性を阻害する仕組みだ。
画像2はインフルエンザウイルス・ノイラミニダーゼタンパク質とタミフル(黄色の分子)の結合系の立体構造(PDB-ID:2HU4)。赤色と青色はそれぞれFMO4-MP2/6-31G計算で得られたタミフルと、各アミノ酸残基との引力的あるいは斥力的相互作用を表わし、色の濃さは相互作用エネルギーの大きさに対応する。なお、この図ではタミフルは単一のフラグメントとして計算された。
この相互作用を計算する場合、従来法ではタミフル全体をひとまとめとした解析しか行えず、ドラッグデザインに必要な、官能基単位の情報が得られないという状況だった。
しかし、今回の手法ならタミフルを4つの機能部位に分割することができるので、NAとの相互作用を機能部位ごとに詳細に解析することが可能となる(画像3)。FMO4計算によって膨大な数値データが出力されるという一見するとデメリットはあるが、BioStationソフトウェア群の1つである可視化・解析ツール「BioStation Viewer」を使えば、可視化インタフェースによって利用者は容易に解析作業を進めていくことが可能なので、問題ないというわけだ。
今回の成果は、SBDD指向の新薬研究開発に対してFMO4-MP2による高分解能の解析手法をスーパーコンピュータを利用した超高速処理性と共に提供するものである。また、タンパク質側をリガンド周辺の「主要相互作用部位(ファーマコフォア)」に絞り、普及型のCPUを積む小規模のクラスター計算機上での簡易解析において効果的な加速が得られる「コレスキー分解」も併せてサポートした。
なおファーマコフォアとは、薬剤分子(リガンド)と標的タンパク質の結合は、作用部位の官能基間の各種の相互作用によって決まるが、そこで重要な部位であるリガンドおよび相互作用の大きい周辺アミノ残残基の集合に当たるのがファーマコフォアである。
そしてコレスキー分解(CDAM)とは、MP2計算で表れる電子間反発積分を近似的に高速評価する方法。クラスター型の計算機で有効な加速が得られる。
また、地球シミュレータ(ES2)、FOCUSスパコン、Blue(CS1000)の3台において、ABINIT-MPを動作させた時の比較も紹介された。地球シミュレータ、FOCUSスパコン、Blueの順で、まずCPUがSX-9/E、Intel Xeon L5640、Intel Xeon X5670。使用ノード数は128ノード(1024コア)、16ノード(192コア)、4ノード(48コア)。1コアのメモリ容量は、1万2000MB、3500MB、3500MB。
そしてFMO4-MP2/6-31Gレベルでの計算時間は、1.4時間、25.7時間、168時間で、残りの2つの地球シミュレータとの計算時間比は、18.6倍、122.9倍となっている。また、FMO4-CDAM-MP2/6-31Gレベルでの計算時間は、1.2時間、17.9時間、75.9時間となっており、地球シミュレータのFMO4-MP2/6-31Gレベルとの計算時間比は、0.9倍、13.0倍、55.1倍となっている。
現在、京速コンピュータ「京」への対応作業も進めており、「京」の利用による電子状態計算に基づいたスパコン創薬が期待できるとしている。