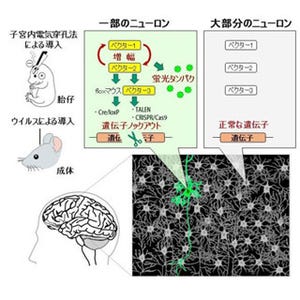
理化学研究所(理研)は11月17日、ゲノム編集ツールであるCRISPR-Cas9システムを利用し、マウス・ラット生体内の神経細胞など非分裂細胞でも有効な新しい遺伝子操作技術「HITI」を開発したと発表した。
同成果は、理化学研究所多細胞システム形成研究センター非対称細胞分裂研究チーム 恒川雄二研究員、松崎文雄チームリーダー、米ソーク生物学研究所 鈴木啓一郎研究員、ベルモンテ教授らの研究グループによるもので、11月16日付けの英国科学誌「Nature」オンライン版に掲載された。
既存のゲノム編集技術の方法では、細胞が活発に分裂する最中に起こるDNA修復機構の一種「相同組換え」修復の仕組みを利用して、ゲノム上の任意の場所に目的の遺伝子を挿入(ノックイン)していた。そのため、皮膚の表皮細胞や腸の上皮細胞などを除くほとんどの生体内の細胞、特に神経細胞や心筋細胞などの細胞分裂をしていない細胞には、この技術を適用することは困難であった。
今回、同研究グループは、非分裂細胞内においても活性を持つDNA修復機構「非相同末端結合経路」に着目。分裂細胞で非相同末端結合経路を利用した遺伝子ノックイン技術の報告は数例あったが、詳細な解析はされておらず、応用できる細胞種の検討も行われていなかったため、今回これらの遺伝子ノックイン法を種々の細胞種で検討した。
また、非相同末端結合経路を利用した遺伝子ノックイン技術に対しては、誤った向きで目的の遺伝子が挿入されてしまうのを防ぐため、導入する遺伝子ベクターの遺伝子配列を工夫し、逆方向の挿入を抑制して安定的に遺伝子を目的の向きで目的の部位に挿入できるよう設計。この方法を「HITI(Homology-Independent Targeted Integration)」と名付けた。
HITIと既存の遺伝子ノックイン方法を比較検討した結果、HITI技術ではヒトの分裂培養細胞において、従来法より約10倍高い効率で遺伝子挿入ができることがわかった。次に、非分裂細胞である生後マウスの神経細胞において、さまざまな遺伝子ノックイン方法を比較・検討したところ、HITIを用いたときのみ高効率で、目的の部位に遺伝子が挿入されたことを確認した。
また、HITI技術をマウス生体に応用し、脳、筋肉、心臓など全身のさまざまな組織や器官で、狙ったゲノム部位に必要な遺伝子を挿入することに成功。さらに、HITI技術を応用することで、網膜の視細胞の変性により進行性の視覚障害をきたす網膜色素変性症ラットモデルの視覚障害を一部回復することにも成功した。
HITIは生体内の一部の臓器で3~10%程度の細胞のゲノムを改変できる技術であり、同研究グループは今後、HITIの分子機構の解明を行うことで技術改良を加え、同技術の遺伝子ノックイン効率の上昇を目指していくとしている。