慶應義塾大学(慶応大)は4月18日、システム生物学の計算モデルを表現するためのXMLベースの記述言語「Systems Biology Markup Language(SBML)」に完全準拠した生化学シミュレータ「LibSBMLSim」の開発に成功し、現在1000種以上あるというSBMLで書かれた生化学反応の数理モデルを利用し、シグナル伝達、代謝、細胞周期、生体リズム、免疫、がんなどの細胞のさまざまな現象のシミュレーションが可能となったと発表した。
成果は、同大 理工学研究科 修士課程2年の瀧沢大夢氏(当時)、同・1年の中村和成氏(当時)、同・2年の田平章人氏(当時)、同・1年の松井達広氏(当時)、同・生命情報学科4年の近原鷹一氏(当時)、同・理工学部の広井賀子専任講師、同・舟橋啓准教授らの研究チームによるもの。研究の詳細な内容は、4月5日付けで英国科学誌「Bioinformatics」に掲載され、LibSBMLSimはオープンソースとして、舟橋准教授の研究室のWebサイトで公開中だ。
近年、生命現象を司る動作メカニズムの理解を目標とした「システム生物学(システムバイオロジー)」的アプローチを採った研究が活況を呈している。システム生物学とは、システム工学の考え方や解析手法などを生物学に応用することで、生命現象をシステムとして理解しようとすることを狙いとした学問である。そして生命現象を司る動作メカニズムを理解するためには、対象とする生命現象に関わる分子の特定、およびそのダイナミクスの理解が必要だ。
一般的にダイナミクスは遺伝子制御、シグナル伝達、代謝など、分子間反応として表現され、ダイナミクスの特性の記述には反応方程式が用いられる。反応方程式の記述は大別して3種類。(1)分子濃度を記述した常微分方程式、(2)分子濃度の空間的分布を記述した偏微分方程式、(3)分子数のゆらぎを考慮した確率モデル、となっている。
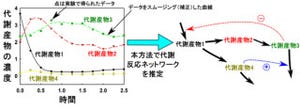
これら異なる記述方法は適用する生命現象に依存する形だが、現在、さまざまな細胞シミュレーション手法が提案されている中で、この10年間において論文などで最も多く利用されているのが、(1)の常微分方程式を用いたシミュレーションだ。ちなみに常微分方程式とはその名からわかる通りに微分方程式の1種であり、未知関数の導関数を含み、独立変数は1つというものである。常微分方程式を用いたシミュレーションでは細胞内の各分子反応を化学反応方程式の形で表し、その濃度変化を1つずつ計算し、未来の予測を行う流れだ(画像1)。
細胞シミュレーションを行う際のシミュレーションしたい現象、例を挙げると、シグナル伝達、代謝、細胞周期、生体リズム、免疫、がんなどだが、それを記述したものを「モデル」もしくは「数理モデル」という。システム生物学の分野では、この数理モデルを記述する言語としてSBMLが普及しており、同言語を利用することで、異なるシミュレータ上で同じ数理モデルのシミュレーションが可能となるというわけだ。例えていうなら、HTML言語で記述されたさまざまなwebページが、さまざまなwebブラウザで閲覧できるのと同じイメージである。
SBML対応シミュレータの開発は活発で、2001年にSBMLが登場して以降、10年強の間に170以上のSBML対応シミュレータが開発されてきた。しかし、SBMLの仕様が膨大であること、生化学反応の数理モデルが複雑であることなどから、SBMLを完全準拠できず、どこかに不具合や欠陥を残したものだった。つまり、SBMLの仕様に完全準拠したシミュレータは実はこれまで存在していなかったのである。
一方で、細胞シミュレーションはこれからの生命科学に必須な技術となってきているのが現状だ。また、SBMLで記述された1000種以上の数理モデルが世界の公的データベースにて公開、また論文と共に出版されており、それらを問題なく利用できる完全準拠のシミュレータが望まれていたのである。
研究チームはこうした現状を踏まえ、SBMLの膨大な仕様に完全準拠するシミュレータの開発を目標としてプロジェクトを開始した。SBMLの仕様に対応するためには、さまざまなシミュレーションエンジン(微分方程式ソルバ)の実装が必須となる。
具体的には、一般的な常微分方程式ソルバに加え、「微分代数方程式」(微分方程式と、未知数に関する多項式で構成される代数方程式の連立方程式)ソルバ、「遅延微分方程式」(現時点の時間導関数が解だけではなく、前の時間の導関数にも依存する可能性があり得る微分方程式)ソルバの実装が行われた。なおソルバとは、高度な数値解析において利用される大規模な連立1次方程式を解くプログラムのことである。
それに加え、シミュレーション中にある条件を満たした場合にのみ発生する「イベント」に対応する必要もあったという。そうしたすべての要件を満たすシミュレータを実装し、正しく動作しているかどうかを全980種のテストケースにて確認が行われ、すべてに合格した(画像2)。こうして、SBMLに完全準拠したシミュレータLibSBMLSimが誕生したというわけである。
またこのシミュレータはライブラリの形式として開発されており、既存のソフトウェアに組み込むことも可能だ。またC、C++、Java、Python、Ruby、C#などのプログラミング言語の関数群を提供しているのでさまざまなプログラミング言語にてシミュレータを開発することまで可能となっている。ライブラリを利用すればわずかなプログラミング、例えばPythonなら3行で独自のシミュレータを開発することが可能で、まさに至れり尽くせりの設計となっている。
生命現象の仕組みを理解するためには、ハードウェアが高性能化している現在は細胞シミュレーション技術は強力なツールとなるのはいうまでもない。ただし、生物学者にとって数理モデルの構築、シミュレータの開発は大きな障壁であることも事実だ。
数理モデルの構築はSBMLの普及により容易になって来てはいるが、これまではSBMLに完全準拠したシミュレータが存在していなかったこともあり、ユーザは数理モデルに応じて使用するシミュレータを使い分けるか、自分の手で開発を行う必要があるという、なかなか手間のかかる状況にあった。
しかし、今回LibSBMLSimが開発されたことで、既存の数多くの数理モデルの正確なシミュレーションを行うことが可能となっただけでなく、このシミュレータをライブラリとして利用することで、わずかなプログラミングで独自のシミュレータを開発することが可能となった。
現在の生命科学では生物学、物理学、化学、数学、計算機科学の融合が必要だという。さまざまな分野の研究者が容易に、かつ正確なシミュレーションを実行できる技術基盤を提供することで、分野融合的な生命科学の研究を促進することが期待されると、研究チームはコメントしている。