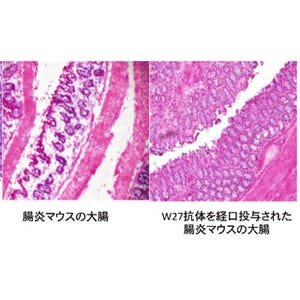
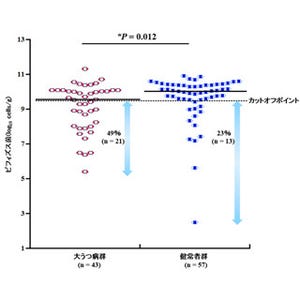
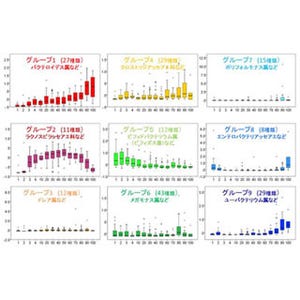
東京大学大学院農学生命科学研究科は、ビフィズス菌がヒトの腸内で糖鎖を分解して定着・増殖するのに関わる酵素の立体構造と機能を明らかにしたと発表した。
同研究は、東京大学大学院農学生命科学研究科の伏信進矢教授、荒川孝俊助教、伊藤佑、佐藤真与、高エネルギー加速器研究機構物質構造科学研究所の千田俊哉教授、松垣直宏准教授、山田悠介助教、Dorothee Liebschner、University of Western AustraliaのKeith A. Stubbs准教授、Mitchell Hattie、Siobhán S. Wills、近畿大学生物理工学部食品安全工学科の芦田久教授らによる研究グループによるもので、同研究成果は、5月25日に雑誌「The Journal of Biological Chemistry」に掲載された。
ビフィズス菌はヒトの腸内に共生し、ヒトの健康に有用な善玉菌としてよく知られている。ビフィズス菌は、栄養源が乏しい消化管の下部に生息するため、ヒトも他の菌も利用できないような分解しにくい構造を持つ難消化性の糖質や食物繊維などを分解して利用するためのユニークな仕組みを有しているという。また、近年、ビフィズス菌はヒトの腸管粘膜の上皮にある糖タンパク質(ムチン)の糖鎖もエネルギー源として生育に利用できることが明らかになった。ムチンに結合している糖鎖は様々な複雑な構造を持っているが、タンパクと結合する根元の部分は、N-アセチルガラクトサミン(GalNAc)という糖がタンパク質中のセリン(Ser)またはスレオニン(Thr)にα-O-グリコシド結合した構造(GalNAcα1-O-Ser/Thr)となっている。
同研究グループは以前より、ヒトの糖鎖をビフィズス菌が利用する仕組みに関する研究を行っていた。今回の研究では特に、ムチン糖鎖の根元において、糖であるN-アセチルガラクトサミンとタンパク質の間の結合を切断する酵素「α-N-アセチルガラクトサミニダーゼ(NagBb)」に着目して研究を行い、α-N-アセチルガラクトサミニダーゼの立体構造をX線結晶構造解析により明らかにすることに成功した。なお、構造決定に際してはS-SAD法という手法が用いられた。同手法では、タンパク質(酵素)を構成するアミノ酸に含まれる硫黄原子の異常分散効果を構造決定に利用するため、通常の構造生物学用放射光実験施設では利用できない、長波長領域の高輝度のX線ビームが必要となる。そのため同研究では、独自に設計・開発された高性能なビームライン(実験施設)であるBL-1AがX線データの測定に用いられた。
α-N-アセチルガラクトサミニダーゼの全体構造や反応中心の構造は、ビフィズス菌が持つ他のファミリーの酵素と部分的に似ている一方で、その活性中心には、糖基質であるN-アセチルガラクトサミンの認識に金属イオンが関与しており、この金属が反応において非常に重要であることが明らかになった。また、この金属は、BL-1Aでの長波長X線を用いた測定実験によってカルシウムイオンであることが推定された。
今回、α-N-アセチルガラクトサミニダーゼの立体構造を解明することにより、ビフィズス菌が腸内の糖鎖を利用して定着・増殖する仕組みがより明確になった。また、ビフィズス菌由来の他のファミリーの酵素との関連や相違点、そして特徴的な金属の関与する反応機構が明らかになったことから、ビフィズス菌はヒトとの共生の過程でこのようなユニークな酵素を進化させてきたことが示唆されるという。同研究は、ヒトと腸内細菌に関する研究において重要な知見を提供しただけでなく、今後、新たなビフィズス菌増殖因子(プレバイオティクス)の開発につながることも期待されるということだ。