理化学研究所(理研)は1月31日、「1分子DNAシーケンサ」を活用した「HeliScopeCAGE法」のサンプル調製工程を自動化して調製期間を従来の実働42日間から8日間まで大幅に短縮し、遺伝子転写開始点を解析する「CAGE(Cap Analysis of Gene Expression)法」の解析スピードを従来の約5倍に向上させることに成功したと発表した。理研オミックス基盤研究領域LSAシステム構築グループ オミックス制御研究ユニットの伊藤昌可ユニットリーダーらによる開発で、成果は米オンライン雑誌「PLoS ONE」に1月30日付けで掲載された。
DNAシーケンサは、DNAを増幅することなく1個のDNAから塩基配列を決定することができる解析装置だ。この装置のDNA塩基配列を高速かつ正確に同定する技術が進歩した結果、ゲノムの大規模解析は遺伝学や医療などのライフサイエンス分野に限らず、環境などほかの分野でも頻繁に使われる研究手法となった。
2011年に研究ユニットは、1個のDNAから塩基配列を同定する1分子DNAシーケンサであるヘリコスバイオサイエンス製「HeliScope」に独自技術であるCAGE法を最適化し、100ngのRNAサンプルから「PCR(Polymerase chain reaction:DNA合成酵素であるDNAポリメラーゼを利用してDNA分子の特定の領域を増幅させる実験手法)」などの増幅なしで遺伝子発現量を定量的に測定するHeliScopeCAGE法を開発している。
CAGE法は、理研オミックス基盤研究領域が開発した方法で、耐熱性逆転写酵素やcap捕捉技術を組み合わせて、5´末端の塩基配列を決定する実験技法のことである。この塩基配列を読み取ってゲノム配列と照らし合わせ、どこからどの位コピーが始まっているかを調べることができる。遺伝子の転写開始点をゲノムワイドに同定できる解析技術だ。
ただしHeliScopeCAGE法には、最新のシーケンサが高速で塩基配列を同定するのに対し、そのサンプル調製には長い時間と手間がかかるという課題があった。そこで研究グループは、解析のさらなるスピードアップとデータの高い再現性を目的に、サンプル調製工程の自動化に取り組んだ次第だ。
HeliScopeでは1回に48サンプルの解析が可能だが、従来の手動技術では、1人が16サンプルを調製するのに7日間を要し、48サンプルを調製するには21日間必要だった。そこで、1度に解析するサンプル数を増やすため、8種類しか調製できなかった8連チューブストリップから96種類を調製できる96穴プレートに変更したのである。
またサンプル調製工程の内、液体サンプルの操作、インキュベーション、サンプル精製の工程を自動化するために、テカン製「Freedom Evo 150プラットフォーム」をもとに、1度に8本の試薬を独立して分注可能なロボットアームを採用。架台には、一定の温度調節が可能な場所と、液体保存部分を備えた。
さらに、RNAを相補的なDNAに変換する「逆転写反応」には複雑な温度調節プログラムが必要となるため、自動開閉加熱ボンネットを装着したプログラム可能な「自動温度調節インキュベーター」を併設した形だ。その上、調製したcDNA(相補的DNA)を定量するための自動計測可能な蛍光プレートリーダーも装備した(画像1)。

|
|
画像1。自動化したサンプル調製プラットフォーム。テカン製Freedom Evo 150をもとに開発。8チャンネル独立分注可能なロボットアーム、温度調節可能部位と液体保存部分を設置した架台、自動開閉加熱ボンネット付きプログラム可能インキュベーター、蛍光プレートリーダーが付属する |
こうして、CAGE法のサンプル調製に必要な全工程(逆転写反応、酸化反応、「ビオチン化反応」、「cap捕捉技術」、核酸の精製操作、cDNAの定量など)を自動化することに成功したというわけである。具体的な工程は、次の通りだ。
- RNAからcDNAを作製する逆転写反応
- キャップ構造のジオール基の酸化反応
- キャップ構造にビオチンを付加するビオチン化反応
- 1本鎖RNA部分を消化するRNase I反応
- ビオチン化されたcap構造を有するRNA/DNA2本鎖を特異的に選別するcap捕捉技術
- 磁気ビーズを用いた核酸精製
- 1本鎖DNAを定量するOliGreenアッセイ
- 品質をチェックする定量PCR
- HeliScopeに供するために必要なポリAテールを付加
手動では一度に16サンプルを7日間で調製するため、48サンプルの調製に実働21日かかるが、自動化工程では一度に96サンプルを8日間で調製できる(画像2)。
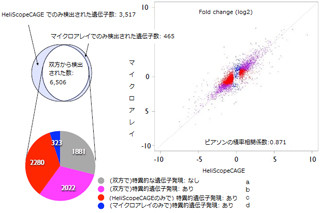
さらに、同じサンプルを自動で調製したものと手動で調製したものを用意し、CAGE解析して比較した結果、転写開始点の位置の同定やその発現量に違いはなく、高い再現性を確認することに成功した(画像4)。
サンプル調製工程の自動化で、1人が96サンプルを8日間で調製できるようになり、手動で要する実働42日間と比べ約5倍効率化したことになる。また、サンプルの取り違えなどヒューマンエラーを未然に防ぐことができ、生産するデータの信頼性向上が期待できるようになった点も大きいというわけだ。
このサンプル調製工程の自動化は、機種が異なる次世代シーケンサに適用したCAGE法へも展開が可能だ。また、RNAの塩基配列を同定する「RNA-seq」や、転写物の全情報を含む「完全長cDNA解析」などの遺伝子解析法にも応用ができるとする。
研究グループでは、今後、さまざまな遺伝子解析のスピードアップとデータの信頼性向上に貢献すると同時に、理研OSCの技術支援サービス「GeNAS」を通じて、外部のさまざまな研究機関へも提供していくとした。