理化学研究所(理研)は3月27日、同研究所が主催する20カ国の114の研究機関の261人の研究者が参加する国際コンソーシアム「FANTOM(FUNCTIONAL ANNOTATION OF THE MAMMALIAN GENOME)」の第5期のプロジェクトとして、正常な細胞を含む各種細胞や組織を収集し、それらのゲノムに存在するゲノムDNAからRNAへの書き写しをコントロールする遺伝子配列の網羅的な解析を実施し、遺伝子近傍にある「プロモータ(遺伝子近位制御部位)」約18万5000個、遺伝子遠方にある「エンハンサ(遺伝子遠位制御部位)」約4万4000個の活性をさまざまな細胞で測定したと発表した。
成果は、理研 予防医療・診断技術開発プログラムの林崎良英プログラムディレクター、川路英哉コーディネーター、理研 ライフサイエンス技術基盤研究センター 機能性ゲノム解析部門のピエロ・カルニンチ部門長、同・センター ゲノム情報解析チームのアリスター・フォレストチームリーダーらが中心的な役割を果たした。
研究の詳細な内容は、3月27日付けで英科学誌「Nature」に主要な2報の論文として掲載された。そのほか、「Nature Biotechnology」、「Genome Research」、「Blood」、「Molecular Biology and Evolution」、「Proceedings of the National Academy of Sciences」、「Nucleic Acids Research」、「Molecular genetics and metabolism」 「BMC Genomics」、「PLoS ONE」の各誌で計18報が発表される。
また、関連する基礎情報は、論文発表と同時に理研のWEBサイトと国立遺伝学研究所の日本DNAデータバンク(DNA Data Bank of Japan:DDBJ)のデータベース上で公開され、バイオサイエンスデータベースセンター(NBDC)にも寄託される形だ。
FANTOMは、ゲノムDNAから転写されているRNAの機能をカタログ化すること(理研のマウスゲノム百科事典プロジェクトで収集された完全長cDNAの機能注釈(アノテーション)を行うこと)を目的として、理研が主催して、林崎プログラムディレクター中心となって2000年に発足された国際コンソーシアムだ。
2011年から始まった第5期(FANTOM5)では、ゲノムDNAに書かれている情報やそれによって制御されるRNAに焦点を当て、細胞機能の解明が目指された。冒頭で述べたプロモータとは、ゲノムDNAがRNAに書き写される時、書き写す領域の先頭部分のゲノム配列近傍にある配列のことをいう。仕組みとして、このプロモータを目印に、「RNAポリメラーゼ」(ヒトの細胞に存在する酵素の1種で、DNAの情報をRNAに書き写す働きをする)が呼び込まれてくるのである。
またエンハンサ領域(配列)とは、遺伝子の転写効率を変化させるDNAの特定配列の内、転写効率を著しく高める部分のことをいう。エンハンサは、プロモータより離れた領域(遺伝子の上流や下流)に位置しており、細胞ごとにゲノムDNAのどの領域をどれだけ書き写すかを規定することにより、細胞の種類(機能)を規定している役割を持つ。しかし、エンハンサの活性を定量する効率的な方法はこれまでに存在しなかったことから、エンハンサ・プロモータの各細胞における活性度合いを明らかにすることが求められていたというわけだ。
そして今回の研究手法における特徴の1つが、がん由来の細胞株だけではなく、正常な初代培養細胞をサンプルとしている点である。世界中から約1000種類のサンプル(初代培養細胞やヒト手術組織、細胞株)が収集され、各々の中で働いている細胞そのままの状態のRNAが精製され、解析に用いられた。この中にはヒトで約400種類知られている正常細胞の内、180種以上が含まれるという。
第2の特徴は「CAGE(Cap Analysis of Gene Expression)法」が活用された点だ。これは、ゲノム上のRNAの書き出し位置を網羅的に同定し、なおかつサンプル中の各RNAの数をカウントすることができる、理研が独自開発した技術である。今回、理研はそれをさらに改良し、「1分子CAGE法」を開発。一般の次世代シーケンサーによる解析ではDNA増幅反応「PCR」が必ず必要となるため、この反応由来の偏りが生じてしまうが、新手法では1分子シーケンサーを用いることで増幅反応を回避でき、測定結果から偏りを少なくできるのが特徴だ。またその検出感度は、数個から10個の細胞中にある1分子のRNAを、99%以上の確率で検出できるほど高いものである。
FANTOM5において、1分子CAGE法による正常細胞の解析が行われた結果、冒頭で述べたように約18万5000個のプロモータが同定され、その内のおおよそ半数が新規発見だったというわけだ。その新規のプロモータは、組織特異的に働いているものが多いのが特徴だという。
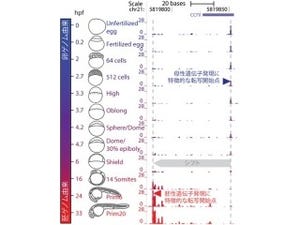
その解析データの1例が下の画像である。横軸はゲノム上の位置で、カウントされたCAGEタグ(ゲノム上の各位置から書き写されているRNA)の数が積み上げグラフとして表示されている。各行は細胞の種類ごとのカウントが示されており、細胞の種類によって活性化している領域が異なることがわかる。
また、多くの細胞のCAGEデータの比較解析をすることにより、こちらも冒頭で述べたように約4万4000個のエンハンサが同定された。なお、これほど大量のエンハンサに関する活性を今回のような多数のサンプルで定量したのは世界でも初めてだとしている。
FANTOM5の今回の成果により、人体を構成する正常な細胞の性質を制御する遺伝子制御部位について、その活性を細胞の種類ごとに測定した包括的データが得られたとする。これは、ゲノムから読み解かれる情報を用いた網羅的かつ体系的な「正常細胞の定義」の基礎となるという。
臨床において病理診断で用いられる組織像などによる細胞の分類には限界があり、細胞の種類を定義づける決定的な方法がこれまでは存在しなかった。今回の研究で得られた細胞の定義を今後さらに充実させることで、ヒトゲノムが生成し得る細胞の全体像が明らかになると期待できるとしている。従って今回の成果は、細胞の多様性がどのように制御されているかという問題の解決だけでなく、病的な状態を定義するために必要な「何が正常なのか」という「正常細胞の定義」の基本になるといえるとした。
また、今回発見されたエンハンサには多数の疾患関連突然変異が発見されたことから、今後、疾患と遺伝子制御の関連の解明に役立つ基礎データとしての利用が期待できるという。
さらに、遺伝子の活動を決定するプロモータやエンハンサを調節する転写因子セットの情報は再生医療、発生・分化を初めとする広範な分野の研究において、基礎・応用の両面から画期的なリソースにもなるとした。例えば今回の成果の中には、iPS細胞を誘導するために必要な転写因子(タンパク質の1種)が結合するプロモータやエンハンサの情報が含まれる。これらの転写因子の情報を用いることで、再生医療などで必要な細胞を、iPS細胞だけでなく、皮膚の細胞などから直接作り出す際に有用なリソースを提供できるようになるとした。
今後、理研では今回の成果を基に予防医療や早期診断医療に資するデータを提供することを目指していくとする。今回の研究では、「正常細胞の定義」を作り出したが、これとがんなどの細胞を比較することで、どのような異常が発生しているのかを詳細に解析することが可能になるという。例えば、「どのようなプロモータ、エンハンサが活性になったか、または活性を失ったか」を調べることにより、がんの悪性度の診断、抗がん剤の有効性、有効な抗がん剤などの評価に貢献すると期待できるとする。
また、各種細胞について定義を作成することは、治療用幹細胞の作成など次世代の再生医療へ向けた重要なステップになる。作りたい細胞で活性化されるべきプロモータやエンハンサを制御している転写因子セットを入手しやすい細胞に入れることで繊維芽細胞からiPS細胞が誘導されるように、多様な細胞を自在に作り出す技術が生まれ、今後の再生医療のスムーズな発展の基礎となると考えられるとした。