理化学研究所(理研)は1月23日、嫌気呼吸を行う好熱性細菌が持つ「一酸化窒素還元酵素(qNOR)」の立体構造解析を行い、好気呼吸酵素に備わっている「プロトンポンプ」の原型にあたる構造を発見したと発表した。この発見は、呼吸酵素が数十億年かけて培ってきた分子進化の道筋の一端を突き止めたことになる。発見は理研放射光科学総合研究センター城生体金属科学研究室の城宜嗣主任研究員と、基幹研究所杉田理論生物化学研究室の杉田有治准主任研究員らを中心とした研究グループによるもので、成果は科学雑誌「Nature Structural & Molecular Biology」に掲載予定で、それに先立ち日本時間1月23日にオンライン版に掲載された。
一酸化窒素還元酵素は、一酸化窒素(NO)を還元し、亜酸化窒素を生成する。「脱窒」(地表に取り込まれた窒素酸化物を再び大気に放出する過程のこと)を行う微生物(脱窒菌)の細胞膜上に存在し、脱窒の途中で産生される細胞毒性が高いNOを速やかに分解する。
NO還元酵素は、生物が生きていくために必須の生理現象である「呼吸」の進化と深い関連があるため、研究者の注目を集めている現状だ。地球誕生後に現れた微生物は、窒素や硫黄の化合物を利用した嫌気呼吸を用いた。しかし、約30億年前に植物の祖先が光合成により水分子から酸素分子(O2)を作り始めたことで、生物の呼吸の様相は劇的に変化。生物はよりエネルギーの高いO2を積極的に呼吸に利用する好気呼吸生物と、O2が持つ細胞毒性から逃れた環境で生きる嫌気呼吸生物に分かれたのである。
好気呼吸では、「シトクロム酸化酵素」が好気呼吸酵素としてプロトン(H+:水素イオン)を利用してO2の還元反応を触媒し、それにより得られるエネルギーを利用して細胞の内側から外側へプロトンを汲み出すプロトンポンプとして機能する。そして細胞膜内外に生じるプロトン濃度勾配によって、生体エネルギーである「アデノシン三リン酸(ATP)」の合成酵素を機能させる仕組みだ。
一方、嫌気呼吸ではNO還元酵素がシトクロム酸化酵素と同様に働き、プロトンを利用してNOを還元し亜酸化窒素を生成する。ただし、シトクロム酸化酵素とは異なり、NO還元酵素にはプロトンポンプ機能はなく、すべてのNO還元酵素は細胞の外側からのプロトンを利用してNO還元を行うと考えられていた。
実際、2010年に研究グループが報告した嫌気呼吸を行う緑膿菌のNO還元酵素(cNOR)の構造には、プロトンを細胞内から外へ汲み出す通り道は存在せず、それまでの定説を支持する結果だったのである。
シトクロム酸化酵素はNO還元酵素から進化する過程で、どのようにプロトンポンプ機能を獲得したのかはこれまで不明で、この疑問を解くために、研究グループは緑膿菌のNO還元酵素とはタイプが異なるqNORの立体構造を詳細に調べることに挑んだ次第だ。
なお、NO還元酵素(NOR)は細胞膜上に存在する膜タンパク質だ。NOを還元し、亜酸化窒素を生成する「2NO+2H++2e-→N2O+H2O」という反応を行う呼吸酵素である。アミノ酸配列および電子供与体の違いから2種類に大別され、細胞膜中に存在する「キノール(quinol)」を電子供与体とするものをqNOR、細胞外の電子伝達タンパク質「シトクロムc」を電子供与体とするものをcNORと呼ぶ。
一般的に、このような膜タンパク質は、溶液中で凝集を起こしやすく構造解析に適した良質な結晶を得ることが困難だ。研究グループは、NO還元酵素の結晶を得るために、高温下で生育し、熱安定性が高く扱いが容易な好熱性細菌「Geobacillus stearothermophilus」が持つqNORに注目。さまざまな精製条件や結晶化条件を検討した。
その結果、良質な結晶を得ることに成功し、微小結晶でも高い分解能で解析できるSPring-8を用いてX線結晶構造解析を行い、qNORの立体構造を2.5Åの分解能で決定することができたというわけである。
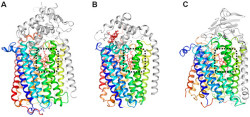
構造解析の結果、qNORの全体構造は、研究グループが2010年に明らかにした緑膿菌のcNORや好気呼吸酵素であるシトクロム酸化酵素と類似していた(画像1)。qNORの活性部位も、cNORとよく似ており、ヘム(有機化合物であるポルフィリン分子の中心に鉄原子が配位した錯体分子)と金属原子から構成され、その近くには酵素機能に必須である2つのグルタミン酸が存在していたのである。
|
|
画像1。呼吸酵素の全体構造の比較。(A)qNOR、(B)cNOR、(C)シトクロム酸化酵素の全体構造。酵素の中心となる部位は(カラーで示している)、すべての呼吸酵素でよく似た構造をしている。また、酵素反応が行われる活性部位(黒色の点線で囲った部位)には、活性や電子伝達に関わるヘム分子(赤色のスティック)が存在する |
ところが、NO還元反応に必要なプロトンの通り道を見比べると、cNORとqNORの間に大きな違いが確認された。cNORでは、細胞の外側から活性部位へとつながるプロトンの通り道が存在していたが、qNORには、対応する位置にプロトンの通り道が見られなかったのである。
その代わりにqNORでは、新たに細胞の内側から活性部位へと続くプロトンの通り道が発見された。この発見したプロトンの通り道の役割を検討するために、アミノ酸置換体を利用した実験や理論化学に基づいた分子動力学計算を行った結果、プロトンを細胞の内側から活性部位へ導きNO還元反応に使われていることが判明したのである。この発見は、「すべてのNO還元酵素は細胞の外側からのプロトンを利用してNO還元を行う」というこれまでの定説を覆すこととなった。
また、大変興味深いことにqNORに存在するプロトンの通り道は、シトクロム酸化酵素のプロトンポンプの一部と類似する位置に存在。具体的には、シトクロム酸化酵素のプロトンポンプは、細胞の内側から活性部位を経由して外側へという道筋になっていたが、qNORのプロトンの通り道は、シトクロム酸化酵素のプロトンポンプ経路のうち、細胞の内側から活性部位へとつながる部分と一致していたのである(画像2・桃色部分)。
以上の結果から、qNORには、シトクロム酸化酵素におけるプロトンポンプの原型ともいえる「試作品」が存在していることが明らかとなり、シトクロム酸化酵素の始原型酵素であると推測するに至ったという次第だ。
今回qNORの立体構造を解明したことで、遺伝子配列の解析から進化的に関連がある2種類の嫌気呼吸酵素(cNORとqNOR)、およびさまざまな種類の好気呼吸酵素(シトクロム酸化酵素)の構造が勢ぞろいした。
これまでは、遺伝情報に基づくアミノ酸の配列比較から呼吸酵素の分子進化に関する予測をしていたが、個々の呼吸酵素の立体構造の比較が可能になり、生物が長年かけて培ってきた分子進化の戦略をより詳しく理解することが可能だ。特に、呼吸酵素の分子進化におけるブレークスルーとなったプロトンポンプ機能の獲得について大きな手がかりを得ることができた形である。
今後は、今までに得られた知見と理論計算による予測を組み合わせて、NO還元酵素にプロトンポンプ機能を付与するなど人工的な機能変換や、還元反応中のNO還元酵素の立体構造解析にも挑戦し、呼吸酵素の分子進化、特にNO還元からO2還元への機能変換の過程について、新たな知見が得られるものと期待しているという。
また、このような知見は、呼吸酵素の分子進化についての理解を深めるだけでなく、タンパク質の高機能を模倣するような人工分子の設計指針にもなりうると期待できると、研究グループでは述べている。