京都大学(京大)iPS細胞研究所(CiRA)は、DNAの脱メチル化を引き起こす候補因子の1つ「Aid(Activation-induced cytidine deaminase)」の検討を実施し、iPS細胞誘導時に必須の役割を持たないことを示す結果を得たと発表した。
同成果は、CiRAの島本廉 研究員、CiRA初期化機構研究部門の沖田圭介 講師らによるもの。詳細は「PLOS ONE」に掲載された。
iPS細胞誘導の際にDNAの脱メチル化が重要であることが報告されているが、そのメカニズムは十分には理解されていなかった。
Aidは、DNAを構成する塩基の1つ「シトシン」がメチル化されたメチル化シトシンを基質とする脱アミノ化酵素で、近年の研究から、ゼブラフィッシュの初期胚においてAidがDNA修復経路を介してDNAの脱メチル化に寄与することが報告されていたほか、マウスの初期胚や脳においてもDNAの脱メチル化に関与することが報告されていた。また、ヒト線維芽細胞をマウスES細胞と融合させて初期化させる場合には、AidがOct3/4のプロモーター領域のDNAの脱メチル化に関わることも報告されており、こうしたことから今回、研究グループは、iPS細胞誘導時のDNAの脱メチル化においてもAidが関わる可能性があると考え、この仮説の検証のためにAidを欠損したNanog-GFPレポーターマウスからiPS細胞を作製し、その性質についての調査を行った。
その結果、Aid欠損マウスと野生型マウスの間にiPS細胞の誘導効率の有意な差は認められないことが確認され、Aidの有無はiPS細胞の誘導効率に影響しないことが判明した。
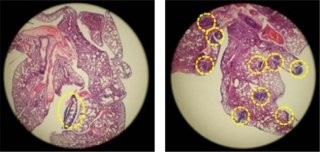
そこで、Aid欠損iPS細胞の性質について調べたところ、Aid欠損iPS細胞の増殖能力は野生型と同等であり、細胞内の遺伝子の発現の解析からも、その違いは僅かであることが判明したという。また、キメラマウスを作製することも出来ることが確認されたとのことで、これらの結果から、Aid欠損iPS細胞は野生型iPS細胞と同等の自己複製能力と分化能力を持つことが示唆されたという。
さらに、Aid欠損iPS細胞のDNAのメチル化領域の解析に向け、MBDシーケンスを行ったところ、Aid欠損型と野生型iPS細胞のDNAのメチル化領域は99.5%が共通していることが判明。Aid欠損はiPS細胞のDNAのメチル化状態に大きく影響しないことが示唆されたとする。
なお研究グループでは、この結果について、Aidとは別の脱メチル化メカニズムが存在することが示唆されたとして、今後のiPS細胞誘導時のDNA脱メチル化のメカニズムの解明に繋がることが期待されるとコメントしている。