東京大学は8月20日、シンガポールゲノムセンターの協力も得て、情報、数理科学、分子生物学の専門家による学際的なチームを作り、真核生物における遺伝子の転写過程の数理モデルを構築することに成功したと発表した。
成果は、東大先端技術研究センターの井原茂男特任教授、同・大田佳宏特任助教、同・児玉龍彦教授、同・和田洋一郎准教授、東大大学院 数理科学研究科の時弘哲治教授、同・坪井俊教授、同・西山了允大学院生(当時)、シンガポールゲノムセンターのYijun Ruan教授らの研究グループによるもの。研究の詳細な内容は、7月10日付けで「Physical Review E」に掲載された。
大多数の生物が生きるためには、細胞がDNAの情報を読み取ってタンパク質を生成する活動が不可欠だ。その工程は、細胞の中のタンパク質が担っている。特に、タンパク質「ポリメレース」がDNA上を走行し、DNA配列の情報を次々と読み取って、タンパク質を作る基になるRNAを産生する」と長年考えられてきた。
この過程はアメーバのような単細胞生物では正しいのだが、複雑な機能を持ったヒトを含む真核生物の転写過程では正しくないことが最近の10年間でわかってきたところだ。
最新の研究では、真核生物の細胞の核の中では、ポリメレースタンパク質が集合していくつかの塊を作り、そこにDNAが入り込んで転写が起きる、という実験結果が得られている(この塊はあたかもRNAを作る工場のように見えるため、「転写ファクトリー」と呼ばれている)。
近年はゲノムプロジェクトにより、遺伝子情報であるDNAの配列についてはかなりの精度でわかってきた。遺伝子情報を取得するための計測技術は各段に進歩し、実験により多くのデータが得られている状況だ。
しかし、真核生物の転写過程は複雑で、現実に見合う数理モデルを構築することは生物の分野で最も難しい問題の1つだった。遺伝子の転写過程を表す数学モデルが整備されていないため、定量的にデータを解釈して生物学的な意味付けをすることが困難だったのである。
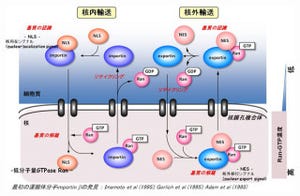
今回構築された数理モデルでは、画像1のように、DNAをある間隔で分割し、ポリメレース(画像中では玉で表現)は隣接する箱と箱の間をある規則で移動すると仮定。
ポリメレースは同じメッシュ(箱)に2つは入れず、遺伝子の端から次の遺伝子まで距離に無関係に、1次元で見た時にある確率pの割合で長いジャンプができるとする。
この時1-pの確率で、ポリメレースは普通にメッシュを次々と移動していくが、DNAがループを作って遺伝子間の距離が近くなった時、ポリメレースが3次元的なジャンプをするとした。
このモデルが正しいかどうかを確かめるため、現実の遺伝子に新たに提案した3次元ジャンプモデルを適用して計算。その結果、ポリメレースが一次元モデルで予測した到達時間よりも早く到達するという最近の実験から得られた現象が、提案したモデルから説明できることがわかった。
このモデルでは、転写が始まるとループの形成が進み、それにつれて確率pが時間と共に増加するので、ポリメレースは時間と共に、遺伝子に長い時間存在するようになり、遺伝子の間で(あるいは、エクソンとエクソンの間で)ジャンプしていくことになる(画像2)。
|
|
|
画像2 ループ構造のある遺伝子位置でポリメレースが運動する様子。ポリメレースを玉で表示、赤が画像1の白の部分、ポリメレースが停留するエクソン位置(黄色)とloopを作るタンパク質の結合位置(緑)を示す |
イントロンの特別な位置にポリメレースが蓄積している時は、その位置とエクソン間でもジャンプが起こることも判明した。これは、道路が混んでいる時に渋滞や遠回りを避けるために、クルマから鉄道など別の交通手段を使ったり別のルートを選択したりすることと同じような振る舞いをしていることを意味している。
なお今回のモデルは、転写ファクトリーが長い時間経った時にどのように振る舞うか、かつその形成過程のメカニズムを説明できるモデルだと考えられるという。
さらにこのモデルで、ポリメレースの数がメッシュの数に比べて変化する時に、ポリメレースの運動がどのように変化するかをシステマティックに計算し、さまざまな遺伝子の振る舞いを予測することにも成功した。
今回の実験では、ポリメレース個々の分子の3次元ジャンプを直接観測することはできていない。ただし、ポリメレースが多く集まる箇所は、DNAループが形成されており、3次元ジャンプが起こり得る程度にDNAが近接していることを分子生物学的な実験によって示すことができた形だ。
以上の結果から、さまざまな状態の細胞の活動を今回提案した新しい転写過程モデルを用いることで、より正確にコンピュータシミュレーションの計算ができ、真核生物特有の転写ファクトリーの形成、機能が予測でできるようになったとした(画像3)。将来的には、新薬の開発や新しい治療方法の開発の期間を短縮し、臨床実験のコストの削減につながることが期待されるという。
画像3は、長い(221.209 kbp)遺伝子であるSAMD4A遺伝子を用いた従来モデルと新モデルによるシミュレーション結果の実験結果との比較。イントロンは赤のバー、エクソンは黄色のバーで示されている。緑のリングはDNAのループ形成に関与するタンパク質「CTCF」が結合している確率が高いDNAの場所を示す。
3つのグラフについてだが、まず(a)は、刺激後の時間t=0(t=30(分)でもほぼ同じ)の時のCTCFが結合している確率が高い遺伝子での位置。および、t=0とt=30(分)後の遺伝子での位置に対するポリメレースの密度を表したもの。
(b)はポリメレースの3次元移動を考えないモデルで計算したt=30(分)後の、CTCFが結合している確率が高い場所でのポリメレースの密度だ。この計算結果は全体的な実験結果をほぼうまく説明するが、RNAの計測実験から同定できるポリメレースの先頭(青玉で表現)がL(黒矢印)に到達した時、P4とP5の位置(赤矢印)でポリメレースの密度が高くなることを説明できない。
(c)は3次元移動を取り入れたモデルで計算したt=30(分)後の、CTCFが結合している確率が高い場所でのポリメレースの密度。CTCFが結合している確率が高い場所である転写開始点からP1、P2、P3、P4にポリメレースの3次元の移動が起こるとすると、P4とP5の位置(赤矢印)でポリメレースの密度が高くなる実験結果をうまく説明できる。なお、3次元のジャンプ確率は等しく0.2だ。
また、確率pの導入によって構造変化を記述できるという今回のモデルは、生物医学以外にも、相転移現象の解明や、物性物理学、情報科学、交通工学、社会学などさまざまな分野にも応用が可能だ。
一般に、ある要素が動くことによって、別の要素からなる構造が大きく変化し、かつ元の要素もより動きやすくなるようなことが、さまざまな現象に見られるが、その状態を記述するモデルを構築する上で役立つことも期待されるとしている。