東海大学などは7月20日、英国Oxford Nanopore Technology製のポータブル型DNAシーケンサ「MinION」に応用可能なゲノム解析システムを開発し、16S rRNA遺伝子をPCR増幅して配列決定することにより、細菌感染症試料に対する細菌の同定を2時間以内で行うことが可能であることを示したと発表した。
同成果は、東海大学医学部基礎医学系 今西規教授らの研究グループによるもので、7月18日付けの英国科学誌「Scientific Reports」に掲載された。
医療機関における細菌感染症の診断は、多くの場合に細菌培養法によって行われているが、培養法による細菌種の判定には1日から2日を要するほか、難培養性細菌の存在や培養条件の不適合などの要因から菌種同定が困難になる場合がある。そうした理由から、細菌感染症が疑われる患者には原因菌の特定を待たずに経験的に選択された抗菌薬を投与するケースが多く、これが有効な薬剤投与の遅れや薬剤耐性菌出現の促進をもたらしていると考えられている。
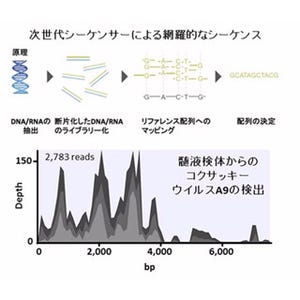
そこで今回、同研究グループは、MinIONとラップトップ型PC2台で構成されるゲノム解析システムを作成した。同システムは、1台目のPCでMinIONを制御し、その出力データを2台目のPCへ連続的にコピーする作業を実行。2台目のPCでDNA配列の詳細な解析を実行するというもの。DNA試料が準備された状態から始めて2時間以内に、DNA配列の読み取りとそこに含まれる主な細菌の同定を行えることが確認されている。また、実際に膿胸患者から得た胸水の検体を解析したところ、原因と考えられる嫌気性菌を迅速に検出することができたという。
ただし、細菌の組成については、独自に開発したゲノムデータベース「GenomeSync」と一般的な相同性探索手法である「BLAST」を用いた方法によって高精度に推定できることが示唆されているものの、計算時間が大幅にかかるため、同研究グループは、さらに改良の余地があるとしている。
従来から特定の細菌を検出するシステムはあったが、同研究グループによると、ゲノム配列データベースに登録されている8万以上の細菌種・系統を特定できる能力を有している点が、従来のシステムとの大きな違いであるという。