京都大学は6月29日、生きたマウスの脳内の細胞内RNAを可視化することに成功したと発表した。
同成果は同大学物質-細胞統合システム拠点(iCeM S)の王丹 特定拠点助教らの研究グループによるもので、6月19日(現地時間)に英科学誌「Nucleic Acids Research」で公開された。
RNAは体内で働くタンパク質が、いつ、どこで、どれだけ必要かという情報を持つほか、細胞内の生体反応の制御を行う役割をもつ。そのため、生体内で目的遺伝子がどのように機能しているかを知るためには、このRNAが細胞内のどこでどのように局在しているのかを突き止めることが必要となる。
RNAが集まり、局在するという現象は正常な細胞でも発生するが、一部の神経変性疾患において異常な集まりがあることが観察されており、この局在変化によって本来のRNAの機能を果たせず、神経細胞の健康状態が破たんしてしまうと考えられている。しかし、生きた組織内でその局在を見るのは困難だったため、RNAの集まりがどのように細胞内で制御され、異常がもたらされているかは明らかになっていなかった。
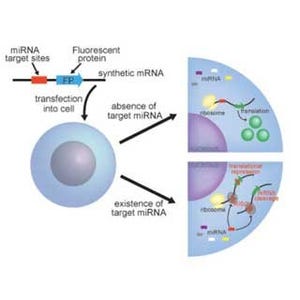
今回の研究では、目的とするRNAの有無によって蛍光のオン・オフができる点灯型蛍光プローブ(DNAやRNAなどの核酸を元にしたオリゴ鎖)を脳内に打ち込み、電流を流して細胞内に導入することで、生きた組織内でのRNAの標識を可能とした。この標識方法を用いて、組織内のRNAの集まりが薬剤に対してどう応答するのか検証したところ、ディッシュ内で培養した細胞と、生きた組織内の細胞では反応に違いがあることを初めて定性的に示すことができた。
同研究グループは「今後は、生きた個体の細胞内でのRNAの集まりが環境応答によってどのように出現・消失するのか、何がそれを制御するのか、正常な組織と疾患にかかった組織でどのように異なっているのかを明らかにすることで、生きた組織・個体での遺伝子発現のメカニズムおよび疾患をもたらすRNAの動きの解明に繋げていきたいと考えています。」とコメントしている。