京都大学は1月29日、サントリー生命科学財団、セルフリーサイエンス、横浜市立大学との共同研究により、抗エイズウイルス活性を有するヒトのタンパク質「APOBEC3G(A3G)」の酵素反応を、「NMR(核磁気共鳴)法」によってリアルタイムで追跡し、定量的に解析することに成功し、A3Gタンパク質がエイズウイルスの遺伝子を効率的に破壊する仕組みを明らかにしたと発表した。
成果は、京大 エネルギー理工学研究所の片平正人教授、同・古川亜矢子 日本学術振興会特別研究員らの研究チームによるもの。研究の詳細な内容は、1月29日付けで独科学誌「Angewandte Chemie International Edition」オンライン版に掲載された。
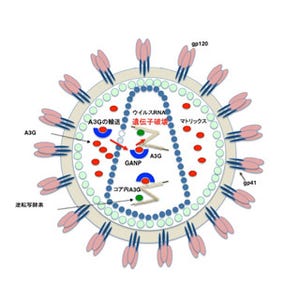
A3Gタンパク質は、エイズウイルスの遺伝子の「シトシン」塩基を脱アミノ化して「ウラシル」塩基に変換することでウイルスの遺伝情報を破壊し、抗エイズウイルス活性を示す(画像1)。
A3GはまずウイルスDNAに非特異的に結合し、その後DNA上をスライディングして標的となるシトシンに到達し、酵素反応を引き起こす。また酵素反応は、ウイルスDNA上の複数の標的シトシンにおいて同時に並行して進行する。「ミカエリス-メンテン法」などの従来の酵素反応の解析手法ではこのような複雑な系を解析することができず、A3Gタンパク質が働く仕組みは謎に包まれていた。
そこで研究チームは今回、まずウイルスのモデルDNAとA3Gタンパク質を試料管に入れ、これをNMR装置にセット。そしてDNA上の複数のシトシンで同時並行に進行する塩基変換反応を、各シトシンに由来するNMRシグナルを別個に観測することで、リアルタイムでモニタリングすることに成功した(画像2)。
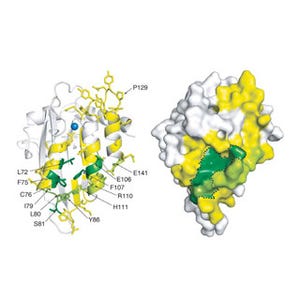
次に、研究チームはA3GのDNAへの非特異的な結合と結合後のDNA上でのスライディングを考慮した反応モデルを構築。そして、画像2で得られた実験結果が定量的に解析された。この結果、A3Gは上流方向にスライディングしながら標的シトシンに到達した際には、下流方向にスライディングしながら到達した際より、脱アミノ化反応の酵素活性が高いことが判明したのである(画像3)。
上流に近い標的シトシンほど脱アミノ化されやすいことはこれまでの観測事実として知られていたが、酵素活性がスライディングの方向に依存するという新たな知見により、このことを合理的に説明することが初めてできるようになったというわけだ。
また脱アミノ化による遺伝情報の破壊がすでになされた直近の部位には、A3Gはもはやあまり作用しないことも確認された。これは、エイズウイルスのDNA上の広い範囲で効率的に遺伝情報の破壊を行うのに適した性質だと考えられるという。このようにNMRシグナルを用いたリアルタイムモニタリングによって、これまで不明であったA3Gタンパク質の動作機構が解明された形だ。
A3Gタンパク質の抗エイズウイルス活性の根幹である脱アミノ化反応の動作機構が解明されたことで、A3Gを活用した抗エイズウイルス薬の創製の活性化が期待されるという。また今回開発されたNMRシグナルを用いた反応のリアルタイムモニタリングと定量解析の手法は、同時並行的に進行するほかの複雑な酵素反応や生体内カスケード反応の解析にも応用可能とした。
またNMRシグナルを用いたリアルタイムモニタリング法は、バイオマスを微生物によって分解することでエネルギーや化成品などの有用物質を獲得する際にも、時々刻々変わる物質群の動態を一網打尽に解析することができ、役立つと考えられるとする。
研究チームは2009年に、A3Gの立体構造の決定にも成功しており、それをA3Gの動的な側面に関する今回の知見と合わせることで、A3Gを活用した新規の抗エイズウイルス薬創製の基盤を確立していきたいと考えているという。またNMRシグナルを用いたリアルタイムモニタリング法を、バイオマスから有用物質を獲得するのに活用することへの利用も開始しており、この進展を目指すとしている。