理化学研究所(理研)と理研のベンチャーであるダナフォームは8月8日、人工核酸を利用した蛍光プローブ「Eprobe(イープローブ)」と「リアルタイムPCR(Polymerase Chain Reaction:ポリメラーゼ連鎖反応)法」を組み合わせることで、特定の遺伝子配列のコピー数や発現量、変異の有無を従来法に比べて正確に検出する方法が開発されたと発表した。
成果は、理研 ライフサイエンス技術基盤研究センター 核酸診断技術開発ユニットの臼井健悟ユニットリーダー、同・マティアス・ハーベス客員研究員、同・花見健志研究員、同・ディアン・デロベル リサーチアソシエイト、理研 予防医療・診断技術開発プログラムの林崎良英プログラムディレクター、ダナフォームのスタッフらの共同研究チームによるもの。研究の詳細な内容は、日本時間8月8日付けで米オンライン科学誌「PLoS ONE」に掲載された。
細胞中における特定の遺伝子のコピー数や発現量を定量的に測定したり、遺伝子型の判別を簡便に検出したりするリアルタイムPCR法は、生命科学研究から遺伝子診断、病原体の検出など臨床分野にまで広く利用されている。リアルタイムPCR法は蛍光プローブを共存させる点が特徴で、反応の進行を蛍光シグナルの増減によってリアルタイムで確認することができる仕組みだ。
この技術の進展には、PCR反応で増幅された鋳型DNA量を検出するための蛍光プローブの開発が寄与してきた。一般的に用いられている蛍光プローブには、PCR反応で生じるDNAの2本鎖に入り込んで蛍光を発する「インターカレーター型」と、配列特異的な検出を可能とする蛍光色素を導入した「人工核酸型」がある。
インターカレーター型の蛍光プローブは検出感度に非常に優れているが、2本鎖DNAであればすべて結合してしまうため、ターゲット以外のDNAも検出し、正確な結果が得られない点が弱点だ。そこで近年、特定のDNA配列を認識する人工核酸型の蛍光プローブを用いて、ターゲットのDNAのみを検出する、新しいリアルタイムPCR法の開発がさまざまな機関で行われている。
これまで報告されている人工核酸型の蛍光プローブは、近接した2個の色素間で励起エネルギーが電子の共鳴により直接移動する現象の「蛍光共鳴エネルギー移動(Fluorescence resonance energy transfer:FRET)」を利用するものだ。しかし、このプローブは構造が複雑で合成が難しいことや、蛍光プローブとDNAの結合による発色反応が非可逆的であるため、増幅したDNA2本鎖を検出できても、その後の1本鎖の解離を検出できないという欠点があった。
リアルタイムPCR法による遺伝子型の判別では、野生型遺伝子と変異型遺伝子でDNAが2本鎖から1本鎖に解離する温度が異なることを利用するため、増幅後に解離した1本鎖DNAの配列を特異的に認識し定量化できる新しい人工核酸型のプローブの開発が求められていたのである。
今回開発されたEprobeは、標的核酸への結合の有無に応じて可逆的な蛍光のオン・オフを行う、かつて理研で岡本晃充博士(現・東京大学教授)によって開発された「励起子相互作用を持つ人工核酸」を利用して蛍光のスイッチングを行う。Eprobeは、DNAの部品であるチミン塩基に蛍光色素(Dye)を2つ持っており、今回使用された蛍光色素は蛍光色が緑色の「D514(チアゾールオレンジ)」と蛍光色が橙色の「D570(チアゾールピンク)」である。
これらの蛍光色素は、ターゲットとなるDNA配列と結合していない1本鎖の状態では、励起子相互作用が働いて蛍光発光が抑制される仕組みだ。EprobeがターゲットのDNA配列と結合し2本鎖を形成すると、2つの色素が分かれてDNAの2本鎖構造に入り込み、色素間の励起子相互作用が解除され蛍光を発するのである(画像1)。
画像1の右にある試験管の上側のものが、励起子相互作用が働いて蛍光発光が抑制されている状態(透明)だ。1本鎖の状態では蛍光を発しないが、ターゲットのDNA配列と2本鎖を形成することによって、下側のように強い蛍光シグナルを示すのである。
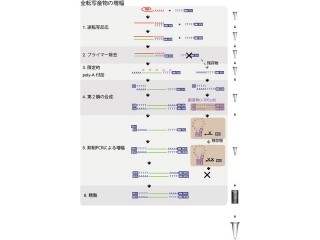
リアルタイムPCRを行う際に十分な量のEprobeを加えておくと、合成直後の2本鎖DNAが加熱によりほどけて1本鎖になった後、温度を下げてプライマーがターゲット配列に結合する「アニーリング」のステップにおいてEprobeも結合し、蛍光シグナルを発する(画像2)。この時発する蛍光を測定することで、増幅DNAの検出を行うというわけだ。また、PCRのDNA伸長過程における温度では、Eprobeが解離できるようにプローブの配列の長さを調整し、DNA合成酵素の伸長反応を阻害しないよう設計されている。
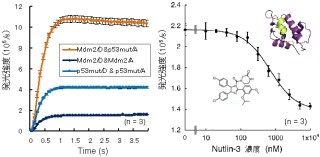
続いて研究チームは、Eprobeを用いたリアルタイムPCR法による定量検出および遺伝子の変異判別を評価するため、ヒトがん細胞に見られる遺伝子変異の領域「EGFR(Epidermal Growth Factor Receptor:上皮成長因子受容体)」を対象にした実験を行った。まず、Eprobeの蛍光に伴う核酸増幅曲線の「Ct値」(増幅が指数関数的に起こる領域で一定の増幅産物量になるサイクル)から算出される定量検出では、102~108コピーまでの広い範囲を検出することに成功(画像3・4)。
次に変異判別の評価のため、野生型遺伝子と変異型遺伝子のそれぞれを増幅したPCR産物に対して、野生型配列をターゲットとするEprobeが解離する時の温度である「Tm値」の解析が行われた。その結果、Eprobeの配列と完全にマッチする野生型のDNA配列ではピークが高温側に、1塩基のミスマッチが生じる変異型の場合にはピークが低温側に出現し、変異の有無を判別することに成功したのである(画像5・6)。
これらの結果から、単一のEprobeを用いたリアルタイムPCR法によって、検出対象となるDNAの定量検出に加え、遺伝子変異の有無を同時に達成できることが明らかとなった。画像3~6のグラフは、Eprobeを用いたリアルタイムPCRでの増幅の定量検出と融解曲線解析を表したものだ。
さらに、Eprobeが、同じ反応チューブでの1回のPCR反応で、異なるプライマーのセットを同時に用いて複数の遺伝子を増幅する「マルチプレックスPCR」に応用できるかどうかの検証も行われた。異なる2つの遺伝子配列をターゲットとし、それぞれ緑と橙色の蛍光を発する色素(D514とD570)で標識したEprobeを用いた実験が行われ、その結果、2つの蛍光色素それぞれの蛍光の増幅と融解に伴うシグナルを同時に検出できることが判明。EprobeがマルチプレックスPCRにも応用できることが示されたのである(画像6・7)。
|
|
|
|
異なる2つの遺伝子配列をターゲットとした、D514とD570の2つの蛍光色素を用いたEprobeを用いたマルチプレックスPCRの結果。(画像6(左))は増幅曲線、(画像7)は融解曲線の解析だ。Eprobeの2つの蛍光色素(D514とD570)、それぞれの増幅曲線および融解曲線を同時に検出できていることがわかる |
|
今回開発されたEprobeは、配列デザインのシンプルさから、これまでのリアルタイムPCRと同じノウハウで遺伝子検査のシステムを開発することが可能だ。この技術を発展させ、ウイルスなどの病原体に由来する低コピー数DNA、RNAの定量的検出、融解曲線解析における1塩基多型(SNP)の遺伝子型判別、がん治療における分子標的薬の患者適合性の診断(投薬前の遺伝子変異検査)などへの応用が期待できるという。今後、Eprobeが臨床での革新的な診断法の開発だけでなく、細胞イメージングと組み合わせることにより生命現象の機能解明を牽引するライフサイエンス技術となることが期待できるとしている。