理化学研究所(理研)と東京大学医学部附属病院は3月28日、日本人集団を対象とした「ゲノムワイド関連解析(GWAS)」を行い、日本人の2型糖尿病の発症に関わる新たな遺伝子領域「ANK1」を発見したと発表した。
成果は、理研ゲノム医科学研究センター内分泌・代謝疾患研究チームの前田士郎チームリーダー、今村美菜子研究員らと、文部科学省「オーダーメイド医療実現化プロジェクト メタボリック・シンドローム関連疾患における個別化医療の実現」実施機関である東大大学院医学系研究科/東大医学部附属病院の門脇孝教授らの共同研究グループによるもの。研究の詳細な内容は、英科学雑誌「Human Molecular Genetics」に掲載されるに先立ち、オンライン版に日本時間3月28日付けで掲載された。
糖尿病患者数は全世界で3億人を超え、2030年には5億人に達するとされている。また、日本の糖尿病患者数は1000万人を超えており、成人の糖尿病有病率は11.2%にも上っている状況だ(出典:国際糖尿病連合「糖尿病アトラス第5版」2011)。
糖尿病患者のおよそ9割は2型糖尿病が占めている。2型糖尿病の発症には遺伝要因が関与しており、今までに国内外の研究によって約50の関連遺伝子領域が見つかっているが、多くは欧米人を対象とした解析で発見されたものだ。
欧米人の2型糖尿病患者では肥満の関与が大きいのに対して、日本人を初めとした東アジア人の2型糖尿病患者では肥満の程度は軽いことが知られており、人種によって2型糖尿病を引き起こす仕組みが異なっていると考えられている。
実際、欧米人対象の解析で見つかった遺伝子領域の中には、日本人の2型糖尿病との関連が見られないものもあることが確認済みだ。このため、日本人における2型糖尿病の遺伝要因を解明するためには、日本人を対象とした解析を行う必要がある。
2010年に研究グループは、日本人における2型糖尿病の関連遺伝子を見出すため、オーダーメイド医療実現化プロジェクトで収集した2型糖尿病患者4470人と対照群3071人のヒトゲノム全体に分布する約50万個の「一塩基多型(SNP=スニップ)」の遺伝子型を基に、ゲノムワイド関連解析を実施。結果、2型糖尿病と強い関連がある「UBE2E2」と「C2CD4A-C2CD4B」の2領域を発見している。
なお、ヒトの染色体にある全DNA情報(ヒトゲノム)は30億にもおよぶ文字の並び(塩基配列)で構成されているが、その99.9%は全人類で共通だが0.1%程度に個人差(遺伝子多型)のあることが判明しており、遺伝情報の1つだけ文字の並びが異なっているもののことを一塩基多型と呼ぶ。30億塩基の並びの中におよそ1000万カ所あり、一部の違いはさまざまな病気のかかりやすさや、薬の効き具合などに関係していると考えられている。
そしてゲノムワイド関連解析とは、病気を発症している集団と、発症していない集団との間で、遺伝子多型の頻度に差があるかどうかを統計的に検定して調べる手法のこと。ヒトゲノム全体を網羅するような数100万カ所のSNPを用いて、ゲノム全体から病気と関連する領域・遺伝子を同定するのである。
今回は、2010年の研究で得られた日本人の2型糖尿病に関連する約50万個のSNPの遺伝子型の情報を基にして、さらなる新規関連遺伝子領域の探索に挑んだ次第だ。
研究グループは、前回の研究で得られた日本人の2型糖尿病に関連する約50万個のSNPの遺伝子型を基に「ジェノタイプインピュテーション(遺伝子型予測)」と呼ばれる手法(画像1)を用いて、解析対象のSNPを約220万個に増やし、新規の2型糖尿病関連遺伝子領域の検索が行われた。
ジェノタイプインピュテーションとは、実際に解析した数10万SNPの遺伝子型情報と、公共データベースに登録されている数100万のSNP情報を基に、専用の遺伝子型予測ソフト(IMPUTE、MACHなど)を用いて、実際に解析したSNPの近くにあるSNPの1人1人の遺伝子型を統計学的に推定する手法のこと。さらにGWASを専用ソフト(SNPTEST、MACH2DATなど)で行い、解析するSNP数を数100万に増やすことが可能だ(画像1)。
GWASで解析した約220万個のSNPの中から、今までに2型糖尿病との関連が知られていない有力な28遺伝子領域にある30個のSNPを選び、さらに別の日本人のサンプル(2型糖尿病患者7605人、対照群3534人)を追加して検証した結果、「ゲノムワイド水準(p=5×10-8)」を超えた2型糖尿病との強い関連を持つ2つの領域(ANK1、MGC21675)が発見された。
ANK1はこれまでに報告のない新規の領域であり、MGC21675領域は、2011年12月に東アジア人集団を用いた解析で2型糖尿病との強い関連が報告された「MAEA領域」と同一であることが判明した(記事はこちら)(画像2、3)。
なおゲノムワイド水準(p=5×10-8)について補足すると、通常の解析の有意水準は、0.05未満を使用する。これは関係がない確率が5%未満という意味であり、100個のSNPを調べるとまったく関係がなくても偶然に5個(5%)は関係があると誤って判断されてしまう可能性があるということだ(偽陽性)。
そこでゲノムワイド関連解析では、通常の判定基準である0.05をさらに100万で割った5×10-8未満という厳しい判定基準を採用して、誤った判断をしないように独自に水準を設定しているというわけだ。
画像2は、ジェノタイプインピュテーションを利用したGWASの流れを図示したものだ。ステージ1では、以前に得られていた日本人の2型糖尿病に関連する約50万個のSNP(患者4470人と対照群3071人)を基にジェノタイプインピュテーションを行うことで、解析対象のSNPを約220万個に増やした。それらをGWASで解析し、今までに2型糖尿病との関連が知られていない有力な28領域のSNP30個が選ばれた形だ。
ステージ2では、別の日本人のサンプル(2型糖尿病患者7605人、対照群3534人)で検証した結果、ゲノムワイド水準(p=5×10-8)を超えた2型糖尿病との強い関連を持つANK1とMGC21675の領域を発見したというわけだ。
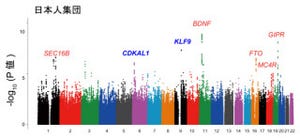
画像3は各SNPと2型糖尿病との関連を調べるGWASの結果の一部を抜粋したもの。その見方だが、横軸にヒトゲノム染色体上の位置、縦軸に各SNPのp値(偶然にそのようなことが生じる確率)が示されている。グラフの上にあるほど関連が高い。青文字の遺伝子領域はこれまでに日本人集団を用いたGWASで同定されている遺伝子領域で、赤文字のANK領域が、今回新規に発見された遺伝子領域だ。
さらに詳細に調べたところ、ANK1領域にある特定の遺伝子型を1つ持つと、1つも持たない人に比べて2型糖尿病発症のリスクが約1.2倍、2つ持つと1.4倍になると推定されることも判明した。
またANK1領域について、欧米の研究グループとの共同研究によって欧米人2万2570人を対象に2型糖尿病との関連について調べたところ、欧米人においても2型糖尿病との関連が認められた(画像4)。
画像4の見方だが、日本人の患者1万6902人を解析した結果、2型糖尿病との関連(p値)は1.37×10-8で、オッズ比は1.18だった。また、欧米人の2万2570人を解析した結果、2型糖尿病との関連(p値)は8.54×10-4で、オッズ比は1.09。この結果より、ANK1領域は、欧米人にも関連があることが判明したというわけだ。なおオッズ比とは、ある集団での疾患リスクをあらわす指標の1つで、オッズ比が2であると危険がおよそ2倍になると推察される。
今回発見したANK1領域がどのようにして2型糖尿病発症に関わるか、そのメカニズムを解明することで糖尿病に対する新しい治療薬の開発に貢献できる可能性があると、研究グループはコメント。
また、今回の成果と、今までに発見された糖尿病に関連する遺伝子多型を組み合わせることで、糖尿病になりやすい人を予測し、積極的な予防対策を講じることによって、糖尿病予防のための生活指導などが可能になってくるものと期待できるとも述べている。